RNA結合蛋白與RNA的相互作用,對于研究細胞內基因調控機制不可或缺。結構學能夠在原子水平上研究分子間的相互作用,而與此互補的方法,比如表面等離子共振(SPR)和等溫滴定量熱法(ITC)能夠用于定量分子互作的親和性以及評估點突變對這些互作帶來的影響。
switchSENSE是一種可以用于測量蛋白與不同種類分子(DNA,蛋白/肽或小分子)間互作的新興技術。在本文的研究中,我們證明了該技術可用于靈活且精確的研究RNA結合蛋白(RBPs)。我們成功地測量出三種RNA結合蛋白(Fox-1, SRSF1和Tra2-β1)與其對應的RNA結合序列之間的互作狀態,并獲得與SPR或ITC測量結果較為接近的KD值。研究結果表明,switchSENSE技術可作為一種替代的方法,研究蛋白-RNA之間的相互作用,可測得的KD值范圍在uM(10-6 ) 到nM(10 -7 to 10-9 ) ,甚至可達到pM( (10 -10 to 10-12 )范圍。底物分子無需進行標記以及低的樣本消耗(蛋白和RNA)使switchSENSE技術較其他技術具有一定的優勢。
RNA是一種多功能分子,除了作為DNA和蛋白之間的連接橋梁,在基因表達方面也發揮著多種關鍵的作用。作為一種短鏈分子(例如miRNA, piRNA, siRNA, snoRNA或snRNA),它能夠形成RNA-RNA雙螺旋,涉及到轉錄后調節過程;作為一種相對更長的分子,RNA能夠形成不同的二級或三級結構,具有酶性質的催化活性(例如核糖酶)或招募RNA結合蛋白到其發揮功能的細胞區域。最后,RNA分子通常受到轉錄后的修飾(例如假尿嘧啶化,2 ' - o -甲基化或堿基編輯),這使得對其活性的調節變得更加復雜。
蛋白質與 RNA 的相互作用在很大程度上被用于調控幾乎所有與基因表達相關的關鍵細胞機制(例如轉錄調節,轉錄后基因沉默或表觀修飾),這也解釋了為什么破壞蛋白與其靶向RNA結合常導致細胞通路的錯誤調控,造成疾病的發生。為了更好的理解基因表達調節機制,需要研究蛋白-RNA復合物,發現新的疾病治療策略。因此開發新的技術用于剖析分子間相互作用就顯得十分重要。

對蛋白-RNA復合物的結構分析一般是用于在RBPs行為模式的原子水平上進行解析,作為對結構分析的一種補充,其他的方法,例如ITC或SPR,常用于解析RNA或蛋白分子的突變對于復合物之間親和性的影響,并鑒定對復合物形成最重要的接觸位點。近來,Dynamic Biosensor(DBS,Germany)公司開發了一種稱為switchSENSE的技術,利用生物芯片,可將DNA或感興趣的蛋白固定在該芯片上,用于快速的檢測蛋白-DNA,蛋白-蛋白和蛋白-小分子之間的親和性和動力學(KD,Kon和Koff)。
switchSENSE技術的原理是金屬微電極上短鏈DNA受變換的電壓驅動下的可控性擺動。根據施加于電極上的電壓,DNA鏈被排斥遠離金屬表面或被吸引靠近金屬表面。可通過共價連接在DNA分子末端的熒光分子的熒光變化實時監測DNA的運動過程。與錨定鏈互補的 DNA 鏈可以通過一種配體分子進行功能化處理,這種配體分子理論上可以是任何能夠通過常規化學和生物化學方法進行連接的分子。此外,第二條鏈本身也能夠作為相互作用分析實驗的靶點,這使得能夠使用各種各樣的核酸結構進行實驗。例如,本研究中,一條單鏈RNA分子與錨定鏈雜交,并且以短的突出的單鏈末端作為蛋白結合研究的靶點。
由于DNA納米桿的擺動發生在水相環境(緩沖液)中,動力學摩擦系數是影響擺動速度的關鍵性參數。配體分子與納米桿共價結合將會增加額外的流體阻力,導致擺動速度減小。底物分子與固定配體的結合將進一步減小擺動速度。這種擺動動力學的改變可實時監測,使得能夠通過分析結合動力學研究分子相互作用。并且,納米桿運動速度減小的程度與附著分子的大小相關(大分子產生的流體摩擦力比小分子更大)。這種效應可用于測量與納米桿結合的蛋白的流體動力學直徑以及配體/底物復合物的直徑。
switchSENSE技術特別適用于研究RNA結合蛋白,因為感興趣的RNA能夠被轉錄并與生物芯片上附著的單鏈DNA(ssDNA)進行雜交。對于該研究,我們使用長的RNA序列(大約60nts),該序列的3’端能夠完全與結合在電極上的含有48個堿基的單鏈DNA進行互補配對,5' 部分對應的是一個側翼的非雜合序列,該序列包含一個由 4 個尿嘧啶組成的連接子、與蛋白質結合的基序以及 GAG 轉錄起始基序(請見Fig. 1A)。利用 T7 RNA 聚合酶在體外成功實現了 RNA 的高效轉錄,并且經HPLC純化。我們接下來必須檢測這種純化的RNA是否能夠有效的和芯片上的ssDNA進行互補配對。一旦RNA-DNA復合物形成,便可通過熒光信號的增加檢測RNA的雜交狀態(請見Fig. 1B). 這種檢測基于當對電極施加負壓時,雜交雙鏈相較于非雜交的單鏈DNA更容易被排斥遠離電極(請見Fig. 1B). 我們測試了不同的 RNA 濃度,并觀察到在水或 T40 緩沖液中,當 RNA 濃度為 1 微摩爾時,與單鏈 DNA 的雜交過程是高效且可重復的(請見Fig. 1B)。我們也觀察到當雜交溫度達到37°C時可增加DNA-RNA雙鏈的形成,可能是因為該溫度限制了RNA二級結構的形成。我們也檢測了使用Dynamic Biosensors提供的再生緩沖液進行再生步驟后,雜交的RNA分子能夠完全從電極上去除。
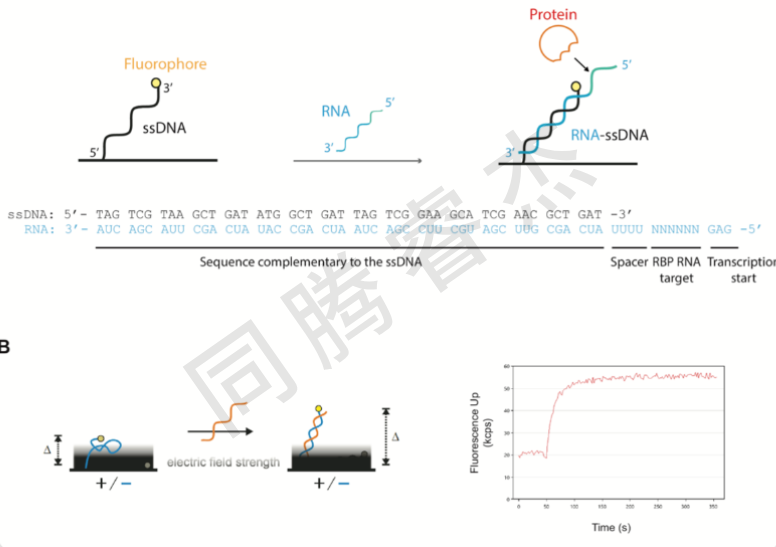
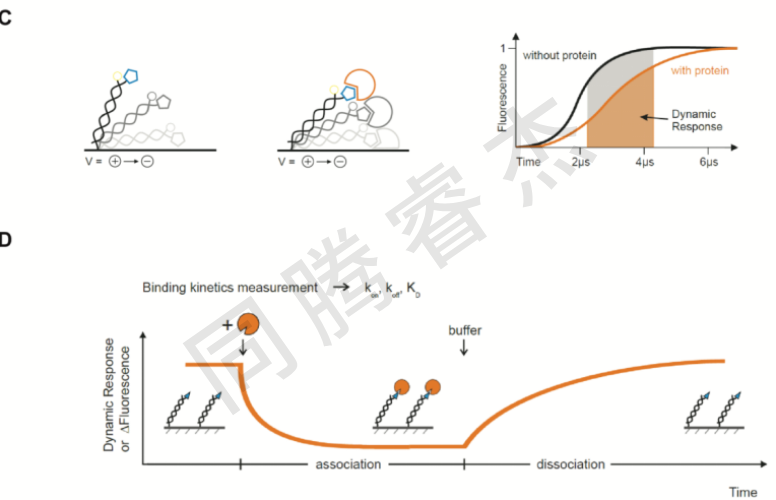
Fig. 1 switchSENSE技術檢測蛋白-RNA相互作用示意圖
我們接著測量結合到感興趣的ssRNA序列側翼上的蛋白是否能夠通過switchSENSE技術被檢測到(請見Fig. 1)。我們決定采用RNA識別基序(RRMs),因為他們能夠在廣泛的親和性范圍內與ssRNA發生互作(KD值從uM到nM范圍)。我們首先測量了Fox-1 RRM, SPR結果已經證明它可以結合到RNA的 5’-UGCAUGU-3’ 序列,KD值為1.09nM。根據該信息,我們轉錄并純化了在5’端非雜交部分包含該序列的RNA分子(請見Fig. 1A),并且通過Dynamic Biosensors的switchBUILD軟件預測了實驗條件。當感興趣的蛋白-RNA復合物沒有相應的KD值的信息時,實驗人員首先需要逐漸增加蛋白濃度,直到觀察到動力學應答熒光信號的改變。估測值通常比能夠觀測到蛋白與RNA結合所需的最小濃度值高10倍左右。使用這個估測的KD值,switchBUILD能夠自動計算用于動力學測量的蛋白濃度和體積,并調節最適流速和結合/解離的時間。通常情況下,該軟件會為待測底物設定一個濃度范圍,使得在與配體結合后的五分鐘內,其信號會有顯著變化。此外,switchBUILD 允許用戶手動調整所有實驗參數。軟件生成的腳本能夠直接在switchCONTROL軟件中打開,該軟件控制著設備。對于動力學測量,我們推薦在每一個解離步驟后移除并雜交新鮮的RNA分子,確保在開始一個新的蛋白結合步驟前,所有結合的蛋白都完全被移除。該蛋白質與目標 RNA 的結合以及隨后的解離過程,通常通過使用不同的蛋白質濃度來進行測定,以獲得準確且可重復的 k(on)、k(off) 和 K(D) 值。
switchSENSE技術的優勢之一是可以通過兩種獨立的方法獲取動力學參數。一個是通過與電極結合的ssDNA 3’端的熒光分子(請見Fig. 1A)。一旦蛋白與雜交的RNA分子結合,熒光信號發生淬滅或增加,在蛋白結合和解離期間能夠實時檢測熒光信號變化。這種方法特別適用于研究兩個大小差異較大的分子間的相互作用(例如蛋白和小分子的互作)。同時,一旦蛋白發生結合和解離,能夠通過檢測DNA-RNA復合物的擺動速度獲取動力學參數(請見Fig. 1C)。蛋白和RNA結合增加了流體動力學摩擦,降低了復合物的運動速度,解離效應與此相反(請見Fig. 1D)。為了確定 DNA 的擺動速度,會持續從對 DNA 運動的觀察中提取動態響應(DR)值,并隨時間進行跟蹤(請見Fig. 1C和D)。雖然在本研究中,我們觀測到的信號改變不足以決定三種蛋白-RNA復合物的動力學參數,但是我們通過追蹤蛋白結合和解離后DNA-RNA復合物的運動速度獲取了高重復性的KD值。因此,這兩種方法根據研究的需要和相互作用性質是互補的。
在本次研究中,我們重點研究三種基于其與 RNA 的親和力差異而形成的蛋白質-RNA 復合物:Fox-1 RRM(KD = 1.09 nM)、SRSF1 RRM2(KD = 0.8 μM)以及 Tra2-β1 RRM(KD = 2.25 μM)。Fox-1 RRM的親和力先前已由SPR測定,我們首選需要檢測是否switchSENSE能夠測得相似的KD值。我們使用6種不同的蛋白濃度,并且使用全局單指數擬合模型分析數據(請見Fig. 2A)。我們得到的KD值為1.49nM,與SPR數據有較好的一致性,但Kon和Koff的數值是不同的,由于在分析數據中使用了不同的擬合模型,使得對速率常數直接進行比較變得復雜:SPR 數據并非遵循簡單的指數時間變化規律,而是需要采用一種復雜的質量運輸限制擬合模型來加以描述,該模型包含了用于補償不完全解離的偏移項;而switchSENSE技術能夠使用簡單的單指數模型進行數據擬合,符合1:1的相互作用預期結果。我們通過研究Fox-1 RRM與polyU的結合,也證明了這種相互作用是特異的。如Figure 2B所示,在測試的所有蛋白質濃度條件下,均未檢測到相互作用。我們接下來測試switchSENSE技術是否可用于檢測野生型Fox-1 RRM和涉及RNA結合殘基發生突變的蛋白之間親和性的微小差異。我們測試了Fox-1 RRM F126H的動力學,其經SPR檢測(KD值為22.1nM),switchSENSE測得相似的KD值(25.3 nM)(請見Fig. 2C)。
在另一項研究中,我們甚至能夠可靠性的檢測到野生型和Fox-1突變型蛋白之間2到5倍的親和性差異(突變蛋白分別為Fox-1 S122A和S155A)。最后,我們測試使用DBS提供的試劑盒將Fox-1 RRM直接偶聯到生物芯片上的可能性,該試劑盒能夠使半胱氨酸連接到與納米桿互補的ssDNA上。由于在Fox-1 RRM的序列中不存在半胱氨酸,我們對166位的絲氨酸進行了改造,該氨基酸不涉及RNA的結合并且是可溶性暴露的。在這種情況下,我們并未發現當固定化的結合物與 RNA 進行孵育時,其動態反應有顯著變化。然而,當 RNA 結合時,熒光信號明顯增強(請見Fig. 2D),其KD值與Fig. 2A中測得的值非常接近(分別是4.39nM和1.49nM)。但兩個實驗中所獲得的Kon和Koff值是存在微小的差異的,這種差異似乎源于蛋白的固定。
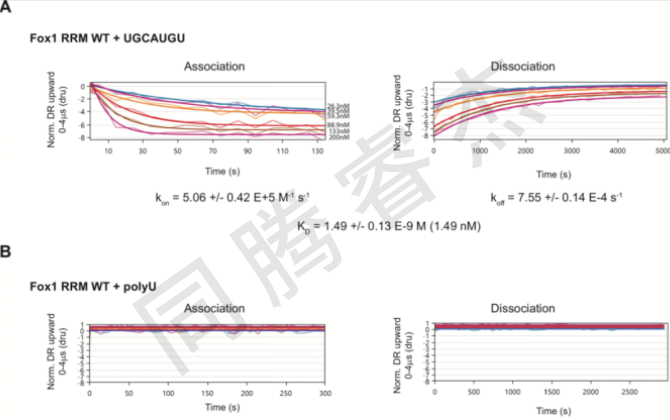
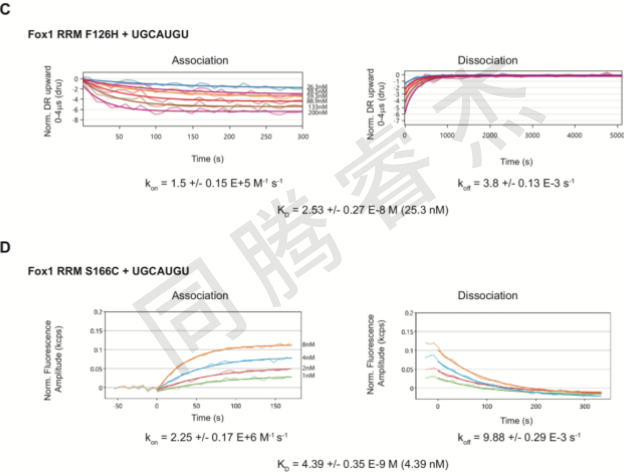
Fig. 2 switchSENSE技術檢測Fox-1 RRM和RNA的相互作用
我們接著測試switchSENSE是否可適用于更高的KD值檢測。我們測量了SRSF1 RRM2-RNA復合物的相互作用,該蛋白的一個結構域通過非經典的識別模式與5’-GGA-3’ RNA基序發生互作,在 5’-UGAAGGAC-3’RNA存在下,ITC測得的KD值為0.8uM。
在本研究中,我們分別測量不同的蛋白濃度,并且所有濃度下的數據都呈現較好的全局擬合。我們測得的KD值比預期的值略低(0.39uM)(請見Fig. 3A)。與Fox-1 RRM相似,陰性對照中未檢測到相互作用(請見Fig. 3B)。
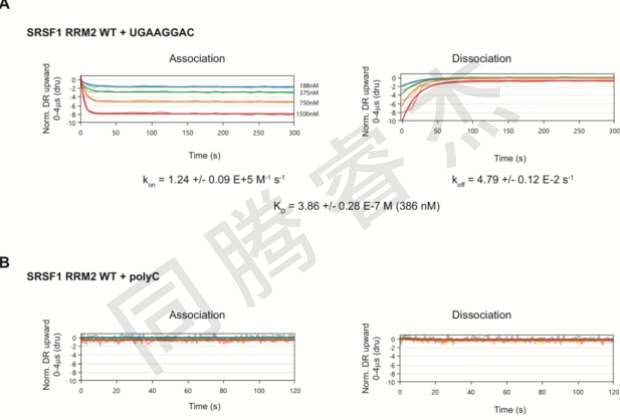
Fig. 3 switchSENSE技術檢測SRSF1 RR2和RNA的相互作用
最后,我們測試在更廣的uM范圍內,switchSENCE是否能夠檢測蛋白-RNA的相互作用。我們重點研究了 Tra2-β1 RRM 這一蛋白,此前我們已解析了其與 5’-AAGAAC-3’ RNA 結合后的結構,并通過 ITC 測定出其 KD 值為 2.25uM。如Fig. 4A所示,即使在該親和范圍下,我們也能夠在6種不同的蛋白濃度下獲得全局性的數據擬合,其KD值為2.89uM。這些蛋白濃度下,未發現與polyU RNA的相互作用(請見Fig. 4B)。最后,我們采用了另一種方法來確定該復合物的 KD 值,即通過報告不同蛋白質濃度下在每次結合步驟結束時測量的 DR 值來實現。這一滴定實驗生成了一張曲線圖,該曲線圖可以使用朗繆爾方程進行擬合。我們得到了一個 KD 值為 1.53 μM(請見Fig. 4C),與通過動力學分析得出的值相近(請見Fig. 4A)。
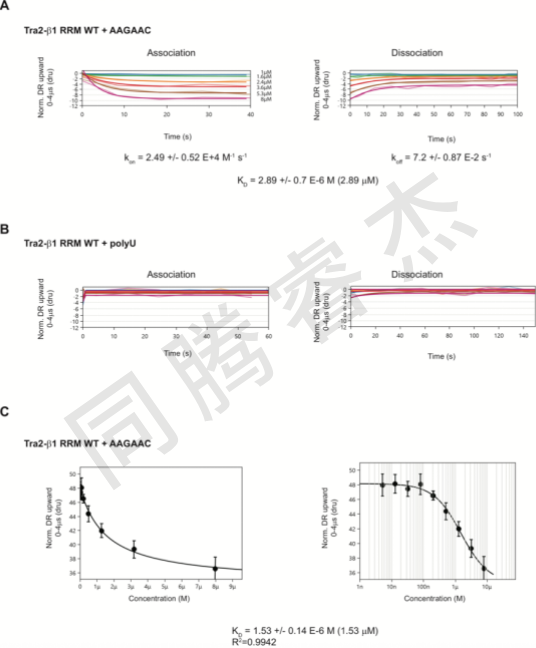
Fig. 4 switchSENSE技術檢測Tra2-β1 RRM和RNA的相互作用




