全長單細胞RNA測序(scRNA-seq)是解析可變剪接、等位基因表達的核心工具,但傳統方法(如Smart-seq3)存在流程耗時(>7小時)、鏈入侵artifacts干擾、難以自動化迷你化等痛點,限制了高通量樣本分析。FLASH-seq(FS)作為革新性全長scRNA-seq技術,通過優化酶促反應與試劑設計,實現測序文庫4.5小時快速構建,還衍生出低擴增版本(FS-LA)和含UMI版本(FS-UMI),靈敏度與基因檢測數顯著優于傳統方法[1]。其中,I.DOT非接觸式納升級移液系統為實驗的高通量與精準性提供關鍵支撐——通過納升級精準分裝試劑,保障384孔板中每孔反應均一性,完美適配自動化流程,推動FLASH-seq在稀有細胞分析、isoform鑒定等場景的落地。
實驗方法
核心平臺與材料設計
采用384孔低吸附板(Eppendorf LoBind),選取HEK293T細胞、人外周血單個核細胞(hPBMCs)、18周人視網膜類器官為研究對象,以Superscript IV逆轉錄酶、KAPA HiFi聚合酶為核心酶試劑,設計三種實驗方案:①FS(標準版,25 μL/5 μL反應體積);②FS-LA(低擴增版,無中間清潔步驟);③FS-UMI(含UMI與5 bp間隔子,減少鏈入侵)。主要檢測指標包括基因檢測數、比對效率、isoform鑒定效率、SNP檢測靈敏度(Figure1a、1e、2a)。
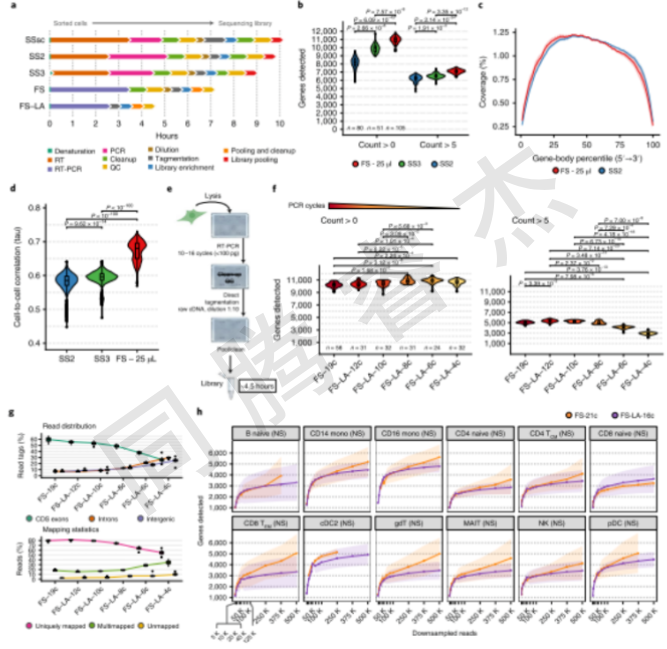
圖1. FS和FS低放大(FS-LA)協議概述。a,為本研究中使用的全長scRNA-seq協議處理96孔HEK293T細胞的估計協議持續時間。步驟用顏色編碼。質量控制包括濃度和粒度分布測量。SSSC,SMART-SEQ單細胞套件(Takara)。b,在兩個讀取閾值下處理SS2(n=80)、SS3(n=51)和FS(n=105)處理的HEK293T細胞中檢測到的基因數量,其中讀取下采樣到500,000(=500K)原始讀取。c,平均值±標準差(S.D.)HEK293T細胞的基因體覆蓋。d,在SS2(n=80)、SS3(n=51)和FS(n=105)中僅使用所有三種方法表達的基因(n基因=20,042)之間的Kendall‘s tau相關性。e,FS-LA工作流。所需的聚合酶鏈式反應周期數是細胞RNA含量的函數。f,使用250K下采樣原始讀取處理的HEK293T細胞中檢測到的基因數量。基因檢測閾值設置為>;0或>;5讀數。g,頂部的面板顯示了用FS或FS-LA處理的HEK293T細胞中映射到外顯子(=CDS外顯子)、內含子或基因間特征的閱讀標簽的百分比,使用ReSQC測量。底部面板顯示了FS和FS-LA的映射統計信息以及唯一映射、多映射或未映射讀取的百分比。h,平均值±S.D.HPBMC樣本中每種細胞類型檢測到的基因數(>;0讀取)。僅顯示兩個或多個單元格支持的點。一些細胞沒有足夠的覆蓋面,無法在每一個點上得到代表。在每種細胞類型的125K讀數下,使用Wilcoxon秩和檢驗(雙側Bonferroni校正,調整P值)來評估FS和FS-LA中基因數量的差異。兩組間差異無統計學意義(P>;0.05)。MAIT,粘膜相關不變T細胞;單核細胞;NK,自然殺傷細胞;Tcm,中央記憶T細胞;TEM,效應記憶T細胞;GDT,γ-增量T細胞;PDC,漿細胞樣樹突狀細胞;cDC2,常規樹突狀細胞2。采用雙側Dunn‘s檢驗,并經Bonferroni校正,調整后的P值為b,d和f。f和g中的盒子圖顯示中位數(中心),第25/75%百分位數(下/上鉸鏈),1.5×四分位數范圍(胡須)和異常值(點)。。
02
關鍵操作與 I.DOT 的核心應用
裂解緩沖液分裝:通過I.DOT將1μL裂解緩沖液(含Triton-X100、dNTPs、FS-dT30VN引物、RNA酶抑制劑等)精準分裝至384孔板,密封后-20℃儲存,保障每孔試劑濃度均一,避免交叉污染(對應文獻Methods“Lysis buffer preparation”)。I.DOT的納升級分配精度(±5%誤差)是后續反應一致性的基礎,尤其適配384孔板的高通量需求。
RT-PCR混合液添加:細胞分選后,從-80℃取出平板,經72℃孵育3分鐘后冰浴,通過I.DOT向每孔添加4 μL RT-PCR混合液(含Superscript IV、KAPA HiFi ReadyMix、甜菜堿等),實現逆轉錄與cDNA預擴增“一步法”(Figure1e FS-LA workflow)。I.DOT的自動化添加避免手動操作誤差,提升實驗重復性,且支持1-384孔靈活適配,契合高通量測序需求。
三種版本實驗流程:①FS:RT-PCR后經磁珠清潔、QC、標簽化;②FS-LA:RT-PCR后直接取1μL cDNA進行標簽化,無清潔步驟;③FS-UMI:RT-PCR混合液中加入含UMI和間隔子的TSO,減少鏈入侵(Figure2a)。
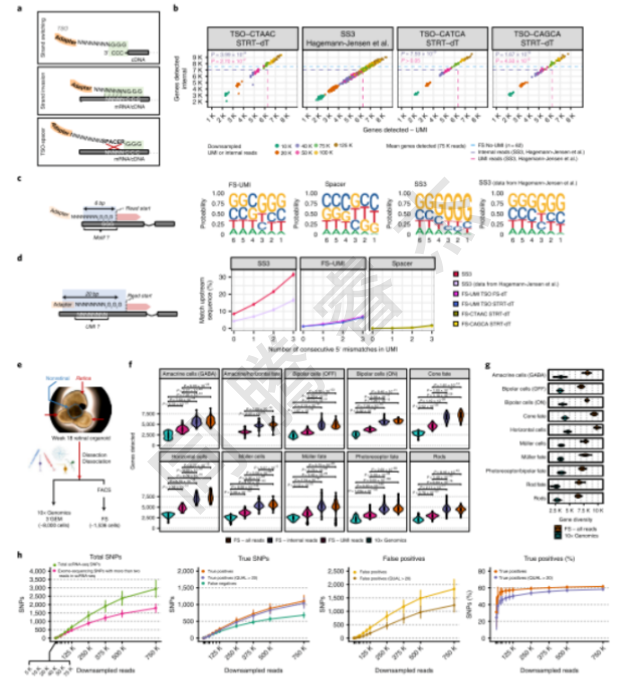
圖2. 在FS-UMI中,鏈入侵事件得到緩解。a,鏈切換反應發生在cDNA末端(上);當UMI和核苷序列相鄰時(中),鏈入侵事件增加,但被間隔區序列減輕(下)。b,使用UMI在HEK293T細胞中檢測到的基因數量與幾個下采樣測序深度(NSS3=85,NSS3_Hagemann-J)的內部讀數之間的關系。=101、NFS-CTAAC_STRT-DT=76、NFS-CAGCA_STRT-DT=16)。藍線表示FS中檢測到的平均基因數(μL=85,75K讀數)。紫色/粉紅色線條表示在SS3中檢測到的基因的平均數量(Hagemann-Jensen等人5,UMI=粉色,INTERNAL=紫色,75K讀數)。比較兩種讀數類型(雙側Wilcoxon秩和檢驗,Bonferroni調整P值,按讀數類型著色)5和其他條件下在75K讀數時檢測到的基因數量。c,使用500K隨機選擇的UMI(HEK293T),在讀出開始附近的6-堿基對中的核苷酸分布。d,均值±S.D.去重復的UMI讀數(%)包含UMI與上游序列之間的匹配,在20個堿基對內,有0到3個連續的5‘不匹配(NSS3=2,089,581,NSS3_Hagemann-J)。=13,511,157,NFS-UMI-TSO_STRT-dt=1,404,599,NFS-CTAAC_STRT-dt=9,964,516,NFS-CAGCA_STRT-dt=1,189,676,UMI讀取)。e,通過寡聚-DT/TSO組合著色。視網膜器質與實驗設計。視網膜和非視網膜部分突出顯示。f,使用10x基因組學和FS-UMI(UMI,內部或兩者同時讀取)在典型細胞類型中檢測到(>;0讀取)基因。Dunn‘s檢驗(雙側,Bonferroni調整P值)。雙極開,開中心雙極電池;雙極關,偏離中心的雙極電池。g、基因多樣性。通過對每種細胞類型的10個細胞重新采樣100次,并計算在兩個以上細胞中使用>;0讀取(包括UMI/內部讀取)表達的基因的數量。h,平均值±S.D.在不同的下采樣讀取深度檢測到SNPs(nsf_retinalods=1,281)。對于每個細胞,轉錄區域的外顯子組SNPs測序(測序深度(=dp)和gt;2)作為參考。在FS-UMI(綠色,DP&>2)或參考(粉色)中檢測到的SNPs總數。在FS-UMI和參照物(橙色,DP>;2;紫色,DP>;2和變異質量(=QUAL)>;20)中檢測到真陽性SNPs。假陰性SNP,FS-UMI中未檢測到參考SNP(深綠色)。假陽性SNPs,在FS-UMI中檢測到,但在參考文獻中不存在(黃色,DP>;2;Brown,DP>;2&QUAL>;20)。真陽性SNPs(%),在參考SNPs中檢測到的真陽性SNPs的百分比。f和g中的框圖顯示了中位數(中心)、25%/75%(下/上鉸鏈)、1.5×四分位數范圍和異常值(點)。
檢測與分析:測序采用Illumina NextSeq 550,數據經STAR比對、featureCounts計數、Seurat聚類分析,評估基因檢測數、isoform多樣性、SNP檢出率(Figure1f-h、2f-h)。
實驗結果
速度與靈敏度雙突破,遠超傳統方法
FLASH-seq全程僅需4.5小時,比Smart-seq3短2-3.5小時(圖1a),且在HEK293T細胞中檢測到的基因數顯著高于Smart-seq2和Smart-seq3(圖1b),即使測序深度降低至500K raw reads,仍能捕獲更多蛋白編碼基因和長鏈基因。FS-LA版本省去中間清潔與QC步驟,上手時間<1小時,且HEK293T細胞10-12個PCR循環即可獲得高質量文庫,hPBMCs僅需14-16個循環(圖1f-g),靈敏度與標準FS相當。
迷你化與自動化適配,I.DOT 助力高通量精準反應
將FS反應體積從25 μL迷你至5 μL后,HEK293T細胞基因檢測數無下降,hPBMCs中CD14?單核細胞、na?ve CD8?T細胞的基因檢測數反而顯著提升。這一迷你化成功得益于I.DOT的納升級精準分配——384孔板中每孔裂解緩沖液與RT-PCR混合液的體積變異系數<3%,避免了手動分裝的誤差,為自動化高通量測序提供了技術支撐。
FS-UMI減少鏈入侵,提升isoform與SNP檢測能力
FS-UMI通過在TSO中加入8nt UMI和5 bp間隔子,顯著降低鏈入侵artifacts:與Smart-seq3相比,其UMIreads中“GGG” motif鄰近比例下降,上游序列匹配率從>10.9%降至<4.25%(圖2c-d)。在250K raw reads下,FS-UMI比Smart-seq3多檢測8±4.3%的基因和18±6.1%的isoform(圖2b),且能在視網膜類器官中檢測到4.1±2.9倍更多的細胞類型標記基因(圖2f)。此外,FS-UMI還能在單細胞水平捕獲SNP,在視網膜類器官中61.57±1.7%的外顯子SNP可被驗證(圖2h)。
多場景應用驗證,適配稀有細胞與復雜樣本
在hPBMCs中,FS成功重構TCR重排,稀有細胞(如MAIT細胞、pDC細胞)的基因檢測數與主要細胞類型相當(SupplementaryFig.3a);在18周視網膜類器官中,FS-UMI比10x Genomics檢測到更多基因和更高基因多樣性(圖2f-g),精準注釋出OFF-center雙極細胞(GRIK1?)、無長突細胞前體(PTF1A?)等稀有細胞類型,還能區分PKM1/PKM2兩種剪接異構體,而10x Genomics無法區分該isoform。
總結與討論
本研究開發的FLASH-seq技術,以“快速、高敏、低artifact”重塑全長scRNA-seq范式,而I.DOT非接觸式納升級移液系統是其實現高通量、自動化、迷你化的關鍵支撐——通過納升級精準分裝裂解緩沖液與RT-PCR混合液,解決了微量反應中體積不均一、交叉污染的痛點,使384孔板高通量測序成為可能,大幅降低實驗成本與上手難度。
技術突破體現在三方面:①速度革新:4.5小時完成文庫構建,FS-LA版本上手時間<1小時,遠超Smart-seq3;②artifact抑制:FS-UMI的間隔子設計減少鏈入侵,提升isoform與SNP檢測準確性;③靈活適配:迷你化至5μl仍保持高靈敏度,兼容hPBMCs、視網膜類器官等復雜樣本,尤其適合稀有細胞分析。
與傳統方法相比,FLASH-seq兼具全長覆蓋優勢與droplet方法的高通量潛力,成本<1美元/細胞,且I.DOT的自動化適配使其易整合到實驗室現有高通量平臺。未來可進一步拓展至臨床樣本快速檢測、單細胞多組學聯合分析等場景,而I.DOT等精準分配技術的應用,也為測序技術的迷你化、自動化發展提供了可復用范式,推動全長scRNA-seq從基礎研究走向臨床轉化。
參考文獻
1、Hahaut V, Pavlinic D, Carbone W, Schuierer S, Balmer P, Quinodoz M, Renner M, Roma G, Cowan CS, Picelli S. Fast and highly sensitive full-length single-cell RNA sequencing using FLASH-seq. Nat Biotechnol. 2022 Oct;40(10):1447-1451.




