微生物單細胞基因組學(SCG)為了解稀有和難培養微生物的基因組提供了手段,是與宏基因組學互補的方法。由于單個微生物細胞中DNA的水平低,因此在進行基因組測序之前需要進行全基因組擴增(WGA)。然而,最常見的WGA方法——多重位點擴增(MDA),十分昂貴且對特定基因組區域有偏差,阻礙了高通量應用,導致基因組覆蓋不均勻。因此,從許多物種(尤其是微生物群落中的少數成員)中獲得高質量的基因組變得困難。在這里,我們提出了一種體積縮減方法,在標準384孔板中顯著降低成本的同時提高了DNA擴增產物的基因組覆蓋度和均勻性。我們的結果表明,為獲得更高質量的微生物基因組,在專業化和復雜的設置(例如微流體芯片)中進行進一步的體積縮減可能不必要。這種體積縮小方法使得SCG在未來的研究中更加可行,從而有助于我們更廣泛地了解環境中那些未被深入研究和未被充分描述的微生物的多樣性和功能。
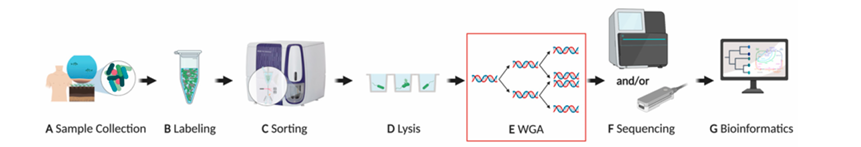
圖1 單細胞基因組學的工作流程。
A:獲得環境樣本后應該立即加工,或在冷凍保護劑的存在下深度冷凍保證細胞的完整性。
B:細胞通常用非特異性熒光染料染色,如 DAPI或SYBR®Green,但它們也可以被特異性標記,例如,熒光原位標記雜化。
C:熒光活化細胞分選(FACS)是最常見的物理分選方法,可將單個細胞分離到多孔板中。
D:一旦單個細胞被分離出來,就會用堿性緩沖液和反復凍融的方法將其破碎,以釋放出細胞中的DNA。
E:由于典型的原核細胞DNA含量低,因此需要進行全基因組擴增(WGA)來產生足夠的DNA用于文庫準備。
F:一旦準備好了DNA文庫,就可以分別使用Illumina®和Oxford Nanopore Technologies®等短讀/長讀測序平臺。
G:利用生物信息學進行序列質量評估、組裝、分類、ORF呼叫和注釋。
一些微生物體內只含有幾飛克DNA,所以其中WGA這一步操作至關重要,為接下來文庫的制備和測序奠定基礎。近幾年來不同的WGA方式也在不斷發展,但基于PCR的擴增方法不適用于DNA含量極低的微生物,因此使用phi29高保真酶的MDA方法應運而生,但MDA整體反應的耗費極高,因此使用受限。在這里,我們列舉了幾種不同的WGA方法。見下表。
表1 微生物單細胞基因組擴增方法的特點
MDA:多重置換擴增;WGA-X:全基因組擴增-X;PTA:原始模板導向擴增;MALBAC:多重退火和循環擴增。
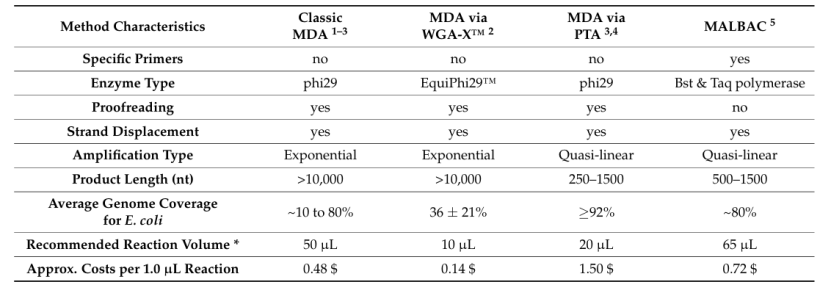
常規的WGA方法反應體系較大,每個反應的花費也較高。因此我們嘗試縮小體系。
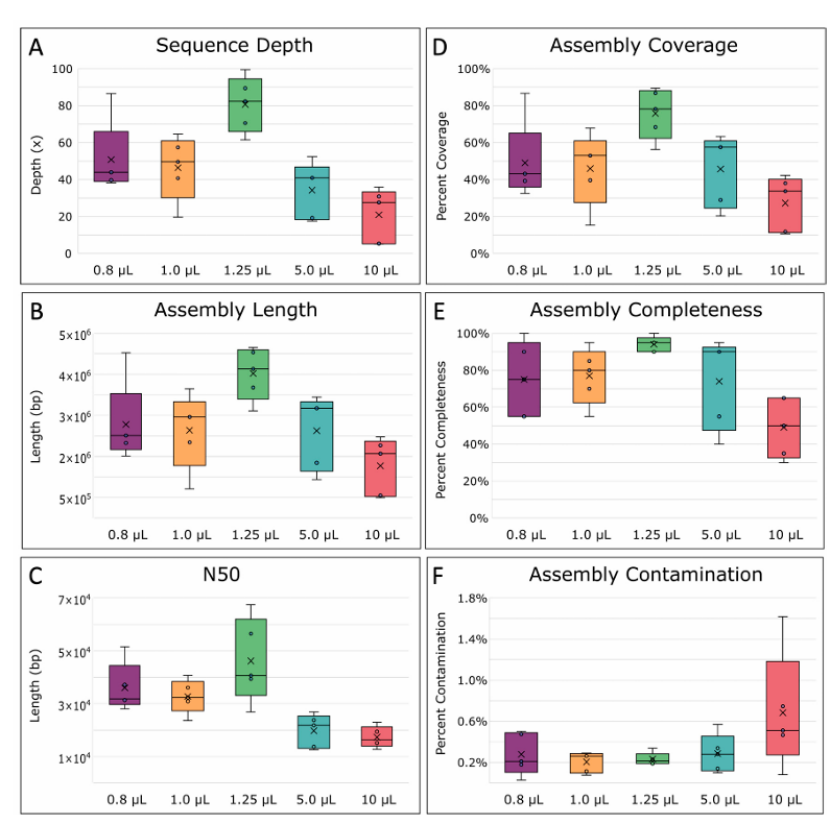
圖2 單倍體基因組(SAG)組裝統計數據。
(A)最終序列深度,即估計的每個基因組堿基平均測序次數。
(B)組裝的總平均長度。
(C)N50平均值,即支持基因組組裝50%的最小片段長度。
(D)組裝覆蓋整個E.oil的百分比。
(E)通過MDMCleaner確定組裝基因組的完整性,
(F)組裝中的污染堿基百分比,方框中間的線表示中位數,x表示平均值。五重復數據計算。
總體而言,這些結果表明,在1.25 uL的反應體積下進行的MDA操作極大地改進了該方法,產生的SAGs相比于標準的大反應體積,具有更少的偏差、更少的污染和更完整的特性。
采用標準384孔板搭配商用細胞分選機和液體分配器,對其他研究人員來說也很方便。標準50 LMDA反應(表1)這種方法降低了97.5%的成本。研究者初步工作發現WGA-X工作1.25 uL反應量也是如此。本文所做的改進將對其他單細胞研究產生極大的影響,從而增加SCG的使用,特別是用于闡明環境樣本中稀有類群和/或新型微生物暗物質的基因組潛力的研究。
在整個工作流程中,分離出單細胞并進行裂解這一步采用了I.DOT非接觸式納升級移液系統。

Dispendix I.DOT非接觸式分液系統作為一款納升級(nl)的微量液體分配設備,采用即時噴點移液技術,系統共有8個獨立控制的正壓通道。進行液體分配時,在底部具有微米級小孔的源板(Source Plate)上方施加一個特定的壓力脈沖,使小孔底部生成一個最小2.55 nl,最大50 nl的液滴,可以釋放到目標板(Target Plate)的任意孔中,每個通道每秒最多可生成100個液滴,通過液滴數量的調節,實現從納升到微升級別的液體分配。
同騰睿杰(上海)生物技有限公司作為CYTENA I.DOT中國總代理商,為您提供優質的售前售后服務。
聯系電話:021-50826962
聯系郵箱:sales@ttbiotech.com




